Le competenze scientifiche del nostro gruppo di ricerca sono rivolte alla genomica applicata e comparata. Eseguiamo assemblaggi e annotazioni di trascrittomi, predizioni di omologia di sequenza e studi filogenetici su sequenze ottenute con metodologie di sequenziamento di nuova generazione.
La grande diversità e variabilità dei geni implicati nell’immunità è alla base della sopravvivenza a lungo termine di popolazioni, specie e individui. L’attività di ricerca del nostro gruppo si concentra sulla genomica comparata del sistema immunitario animale, con particolare attenzione all’immunità innata degli Invertebrati (Bivalvi e Crostacei) e alle strategie che essi mettono in atto per difendersi dall’attacco di patogeni (peptidi antimicrobici, recettori di patogeni, vie di trasduzione del segnale). La nostra esperienza nell'applicazione delle tecniche di biologia molecolare e nella gestione dei dati genomici ha aperto la strada a numerose e durature collaborazioni con altri gruppi di ricerca, su progetti di genomica comparata dei Vertebrati, in particolare dei Pesci. Negli ultimi anni le nostre collaborazioni sono spesso a supporto delle analisi sulla Biodiversità sia animale che vegetale. Quindi applichiamo sia tecnologie di DNA metabarcoding che di DNA ambientale (eDNA).
Genomica dell’immunità dei Bivalvi
(Ricercatori coinvolti: Alberto Pallavicini, Marco Gerdol)
L’evoluzione dei sistemi di difesa innata è alla base dell’eterna lotta tra ospiti animali e microrganismi patogeni. A causa delle ampie variazioni stagionali, le acque costiere sono soggette a cambiamenti considerevoli di temperatura, salinità e contenuto di ossigeno. Tali variazioni possono comportare alterazioni significative delle comunità microbiche e portare, occasionalmente, alla proliferazione di batteri patogeni o alghe potenzialmente nocive. Le acque marine costiere, inoltre, sono tra gli ambienti più colpiti dalle attività antropiche e dal conseguente inquinamento da idrocarburi, metalli pesanti e fertilizzanti.
I molluschi bivalvi filtratori hanno sviluppato specifiche strategie di difesa per far fronte ai potenziali pericoli. Il Mytilus galloprovincialis, per esempio, è particolarmente tollerante nei confronti di una vasta gamma di agenti patogeni e di cambiamenti ambientali e pertanto, viene considerato una specie di riferimento nelle indagini ecotossicologiche. Gli eventi di mortalità causati da agenti infettivi o parassiti sono più rari nelle popolazioni di mitili, rispetto alle altre specie di Bivalvi, tuttavia le basi molecolari di questa resistenza non sono ancora del tutto conosciute. L’argomento assume ulteriore interesse poiché gli Invertebrati non sono in grado di sviluppare risposte efficaci sul lungo periodo, in quanto privi di sistema immunitario acquisito.
Lo scopo principale dei nostri progetti è di comprendere meglio tali meccanismi mediante la combinazione di tecniche di biologia molecolare classica e approcci -omici. L’applicazione delle più recenti tecnologie di sequenziamento massivo ha permesso al nostro gruppo di ricerca di dimostrare che l’espansione delle famiglie geniche implicate e il rapido tasso di mutazione dei recettori e degli effettori immunitari, sono fondamentali per il riconoscimento degli agenti patogeni da parte del mitilo.
Le principali linee di ricerca in materia di immunità dei bivalvi riguardano lo studio approfondito della variabilità e della regolazione dei recettori implicati nel riconoscimento dei patogeni (PRR) e delle vie di trasduzione del segnale coinvolte nella risposta immunitaria contro batteri, virus e altri parassiti. Siamo interessati anche allo studio di molecole effettrici ed in modo particolare dei peptidi antimicrobici (AMP). Abbiamo deciso di utilizzare una strategia alternativa al metodo biochimico classico per l’identificazione di nuovi peptidi bioattivi, ovvero viene eseguito uno screening in silico del trascrittoma per la selezione di sequenze con caratteristiche chimico-fisiche compatibili con attività battericide. Questo approccio ha portato all’identificazione delle miticaline, una nuova famiglia di AMP lineari e cationici espressa nelle branchie, che è attualmente in fase di caratterizzazione funzionale.
Abbiamo recentemente dimostrato, per la prima volta nel regno animale, la presenza di un pan-genoma aperto nel mitilo, caratterizzato da un numero elevatissimo di geni dispensable, soggetti a fenomeni di presenza-assenza genica. Tali geni potrebbero avere un importante ruolo adattativo, spiegando in larga parte l’elevata resilienza di questo bivalve nei confronti di svariati stress abiotici e biotici, nonché la sua invasività.
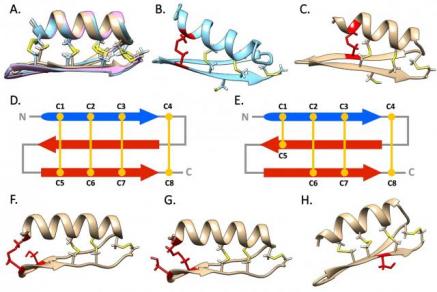
Diversità strutturale di peptidi antimicrobici mytilin-like nei mitili
Genomica comparata ed evolutiva
(Ricercatori coinvolti: Alberto Pallavicini, Marco Gerdol)
Gli organismi non-modello sono stati quasi completamente esclusi dalle analisi genetiche e genomiche, a causa dei costi finora eccessivi per il sequenziamento di genomi così grandi o per la difficoltà di reperire o conservare i campioni in laboratorio. Negli ultimi anni, però, grazie alla crescente accessibilità delle moderne tecnologie di sequenziamento massivo, il nostro gruppo ha effettuato studi su specie di grande importanza nel campo della biologia evolutiva, quali il celacanto ed i dipnoi.

Il celacanto, un “fossile vivente” che si pensava estinto fino alla fine degli anni ’30, ed i pesci polmonati ricoprono difatti una posizione chiave nell’albero evolutivo dei tetrapodi, ed il loro studio può permettere di chiarire molti aspetti tuttora poco compresi dalla transizione dalla vita acquatica a quella terrestre. Le nostre attività in questo ambito comprendono la partecipazione al progetto internazionale di sequenziamento del genoma di Latimeria chalumnae e l’investigazione dei meccanismi molecolari della determinazione del sesso e l’attività degli elementi trasponibili
Più recentemente, il nostro coinvolgimento in un progetto nazionale PRIN nell’ambito della genomica della conservazione ci ha dato l’opportunità di estendere le nostre analisi genomiche a 5 specie endemiche italiane a rischio estinzione, che includono l’iconico orso Marsicano e lo storione dell’Adriatico. L’ottenimento di genomi di riferimento di elevata qualità rappresenterà una valida risorsa per il monitoraggio della diversità genetica delle popolazioni residue, permettendo di studiare molti aspetti ancora scarsamente noti della biologia di questi organismi.
Grazie al coinvolgimento nel Programma Nazionale di Ricerca in Antartide è stato possibile estendere gli studi di genomica comparata a diverse specie Antartiche, interessanti per i loro adattamenti evolutivi ad ambienti estremi. L’utilizzo di approcci trascrittomici per investigare i meccanismi molecolari alla base della tolleranza a temperature polari permetterà di comprendere quali potrebbero essere gli effetti a lungo termine del riscaldamento globale su pesci ed invertebrati bentonici.
Non di minore interesse è lo studio di specie ittiche più comuni nei nostri mari, sia quelle di interesse commerciale che di interesse ecologico. Tra le specie su cui stiamo attivamente lavorando vanno sicuramente ricordati il branzino Dicentrachus labrax, in quanto lo studio molecolare delle complesse risposte immunitarie è di fondamentale importanza per il miglioramento delle pratiche di acquacoltura, ed il pesce abissale Chauliodus sloani, i cui adattamenti fisiologici all’ambiente di profondità sono di grande interesse.
Metabarcoding procariotico ed eucariotico
(Ricercatori coinvolti: Alberto Pallavicini e Fiorella Florian)
La tecnica del metabarcoding permette l’identificazione contemporanea di più specie presenti in un'ampia varietà di campioni quali suolo, acqua, feci, pelle, ecc. Il nostro gruppo di ricerca si è occupato di definire, per esempio, la comunità batterica di bivalvi (mitili, ostriche) in condizioni normali ed in presenza di patogeni che causano moria degli allevamenti. In collaborazione con gli ecologi abbiamo studiato campioni di suolo e di acqua di nicchie marine al fine di valutarne la comunità microbica e zooplanctonica.
Un altro argomento oggetto di studio è il polline e spore fungine presenti in campionamenti d'aria. Per ulteriori informazioni si rimanda al gruppo di ricerca di Biologia della simbiosi lichenica.
Da alcuni anni ci occupiamo del rilevamento e dell'analisi del DNA ambientale (dall'inglese environmental DNA, eDNA) in vari comparti acquatici, quali gli ambienti d'acqua dolce e marini.
Il eDNA è composto da tracce di DNA rilasciate nell'ambiente dalle specie che lo abitano. Con apposite metodiche molecolari, si analizzano queste tracce e siamo così in grado di creare delle mappe di presenza e distribuzione delle specie monitorate. Ad oggi le analisi di eDNA sono state applicate al monitoraggio delle due principali specie di gamberi decapodi nativi della regione FVG a scopi conservazionistici, ma anche per monitorare specie invasive di crostacei e pesci. Per quanto riguarda il comparto marino, il laboratorio è impegnato nella determinazione dello zooplankton marino e nel rilevamento di patogeni emergenti in ambiente marino, come l'Haplosporidium pinnae nella nacchera di mare Pinna nobilis.
Tale metodica ha inoltre aperto una nuova frontiera, l'esplorazione delle interazioni tra uomo e microrganismi. Gli esseri umani, infatti, vivono in costante associazione con i batteri che sono naturalmente presenti all’interno, sulla superficie e nelle cavità del corpo umano e il cui numero è almeno dieci volte superiore a quello delle cellule del corpo stesso. Questo microbioma, è complesso e dinamico ed ha una profonda influenza sulla fisiologia, sulla nutrizione, sull’immunità e sulla salute dell’uomo. Le modificazioni e le alterazioni delle comunità microbiche associate all’uomo sono, infatti, alla base di molte malattie. In tale contesto abbiamo effettuato analisi di metabarcoding microbico su campioni fecali, vaginali e di placca dentale, potenzialmente collegati a diverse condizioni patologiche o di disbiosi.
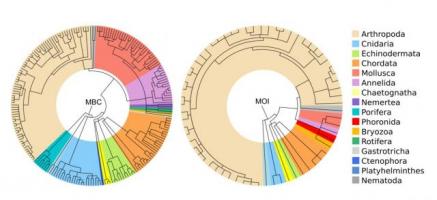
Albero tassonomico rappresentante la ricchezza tassonomica dello zooplancton marino rivelata con metabarcoding (MBC) e ispezione morfologica (MOI)
Maggiori collaboratori Nazionali ed Internazionali negli ultimi 5 anni
- Università di Genova (Paola Canesi, Luigi Vezzulli)
- Università Politecnica delle Marche (Adriana Canapa, Antonio Dell’Anno)
- Istituto Nazionale di Oceanografia e Geofisica - OGS (Paola Del Negro, Federica Cerino, Valentina Tirelli)
- Stazione Zoologica Anton Dohrn - SZN (Sergio Stefanni, Immacolata Castellano, Maria Vittoria Modica, Enrico D’Aniello, Maria Sirakov)
- Università della Tuscia (Giuseppe Scapigliati, Francesco Buonocore, Andrea Miccoli)
- Università di Padova (Paola Venier, Marta Giacomello)
- Università di Ferrara (Giorgio Bertorelle)
- National Institute of Biology (David Stankovic)
- University of Split (Tomislav Roncevic)
- CSIC (Antonio Figueras, Beatriz Novoa)
- Paris Natural History Museum (Nicolas Puillandre)
- University of Ljubljana (Gregor Anderluh)
- Yokohama City University (Yasuhiro Ozeki)
- University of Nagasaki (Yuki Fujii)





